- 新サービス
【新サービス】SLAF-Seq
Specific-Locus Amplified Fragment Sequencing (SLAF-Seq)
大規模集団におけるジェノタイピングは遺伝学的研究の第一ステップであり、機能的遺伝子の探索や進化学的解析のためのベースとなります。このような研究では、サンプルあたりののシークエンシングコストを最小限に抑えて効率的に遺伝子マーカーを探索するため、ディープシーケンシングの代わりにReduced Representation Genome Sequencing (RRGS)が採用されることが多くあります。RRGSは制限酵素でゲノムを消化して得られる特定の長さの断片に注目してゲノムの一部のみをシークエンスします。様々なRRGSの手法がある中で、Specific-Locus Amplified Fragment Sequencing (SLAF)は研究のごとにカスタマイズが可能な卓越した分析手法です。SLAFはプロジェクトごとに使用する制限酵素を最適化することで、反復領域を効果的に避けながらゲノム全体に均一に分布するSLAFタグ(シーケンスされる400-500bpの領域)を作成し、優れた遺伝子マーカーの探索を可能にします。
技術概要
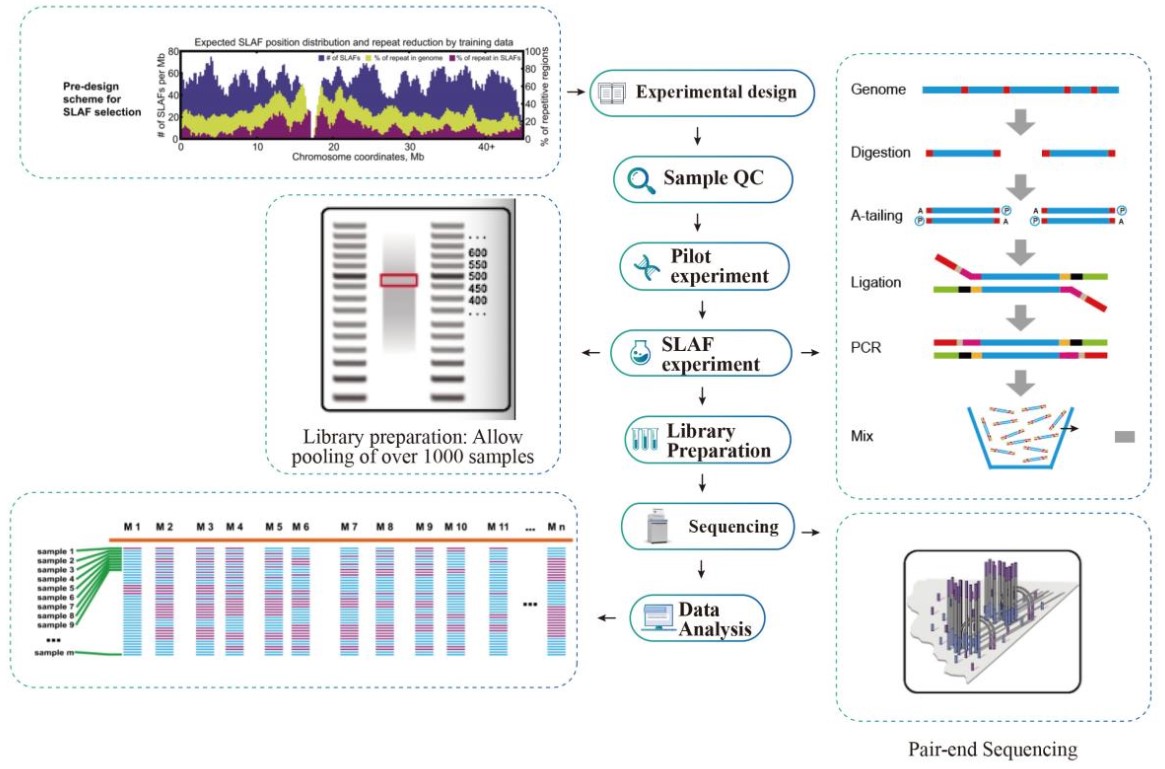
SLAF-Seqサービスの流れと分析の特徴

-ダブルバーコーディングによるライブラリ調製で1000サンプル以上のプーリングが可能
-レファレンスゲノムの有無に関わらず解析可能
・レファレンスあり:SNP/InDelの探索
・レファレンスなし:サンプルのクラスタリングとSNP探索
-in silicoプレデザインで複数の制限酵素の組み合わせをスクリーニングし、ゲノム全体に均一に分布するSLAFタグを探索
-プレ実験では3種類の酵素の組み合わせをテストして9つのSLAFライブラリを作成、最適な制限酵素の組み合わせが選択可能
SLAF-Seqサービスのアドバンテージ
高い遺伝子マーカー探索効率
ハイスループットのダブルバーコーディングシステムを統合することで大規模集団の同時シーケンスを可能とし、部位特異的な増幅が多様な研究プロジェクトの要求を満たします。
ゲノムへの低依存性
レファレンスゲノムの有無に関わらず様々な生物種に適用可能です。
柔軟な実験デザイン
単一酵素から複数の酵素での消化、様々なタイプの酵素を研究目的や生物種に応じて選択することができます。最適な酵素をデザインするためにin silicoでプレデザインを実施します。
高効率な酵素消化
in silicoプレデザインとプレ実験により、染色体上に均一に分布するSLAFタグ(1SLAF tag/4kb)と反復配列(<5%)を考慮して酵素消化を設計します。
SLAF-Seqに特化した独自のバイオインフォマティクス
BMKGENEが開発したSLAF-Seqのための統合的なバイオインフォマティクスのワークフローは納品データの信頼性と正確性を保証します。
技術と知識が豊富な分析チーム
分析チームは植物、哺乳類、鳥類、昆虫、水棲生物を含む様々な生物種で2000以上のSLAF-Seqのプロジェクトの実績があり、あらゆる研究プロジェクトについての豊富な知識があります。
バイオインフォマティクス
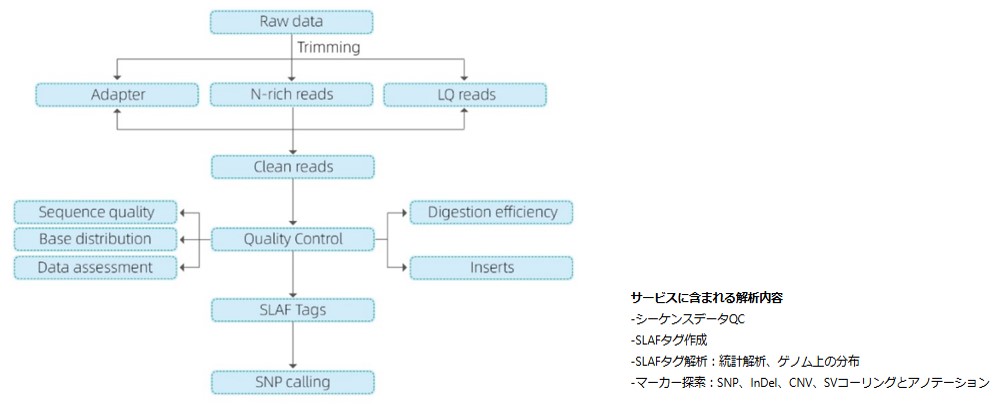
解析データ例
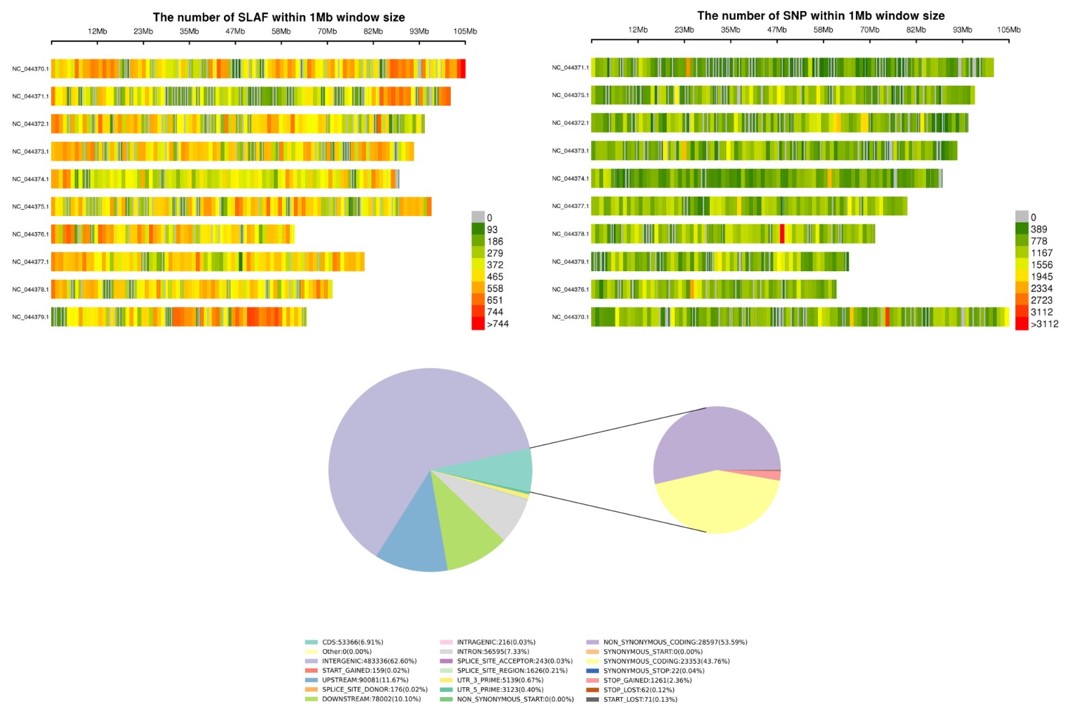
左上:デモサンプルにおける染色体上のSLAFタグの分布
右上:デモサンプルにおける染色体上のSMPの分布
下:SNPアノテーション
お問い合わせ
本サービスはSLAF-Seqを開発した海外ラボにて実施されます。弊社にてお取次ぎをしておりますので、仕様や料金などの詳細はお問い合わせください。
